耐药菌研究流程与多模态应用系统构建
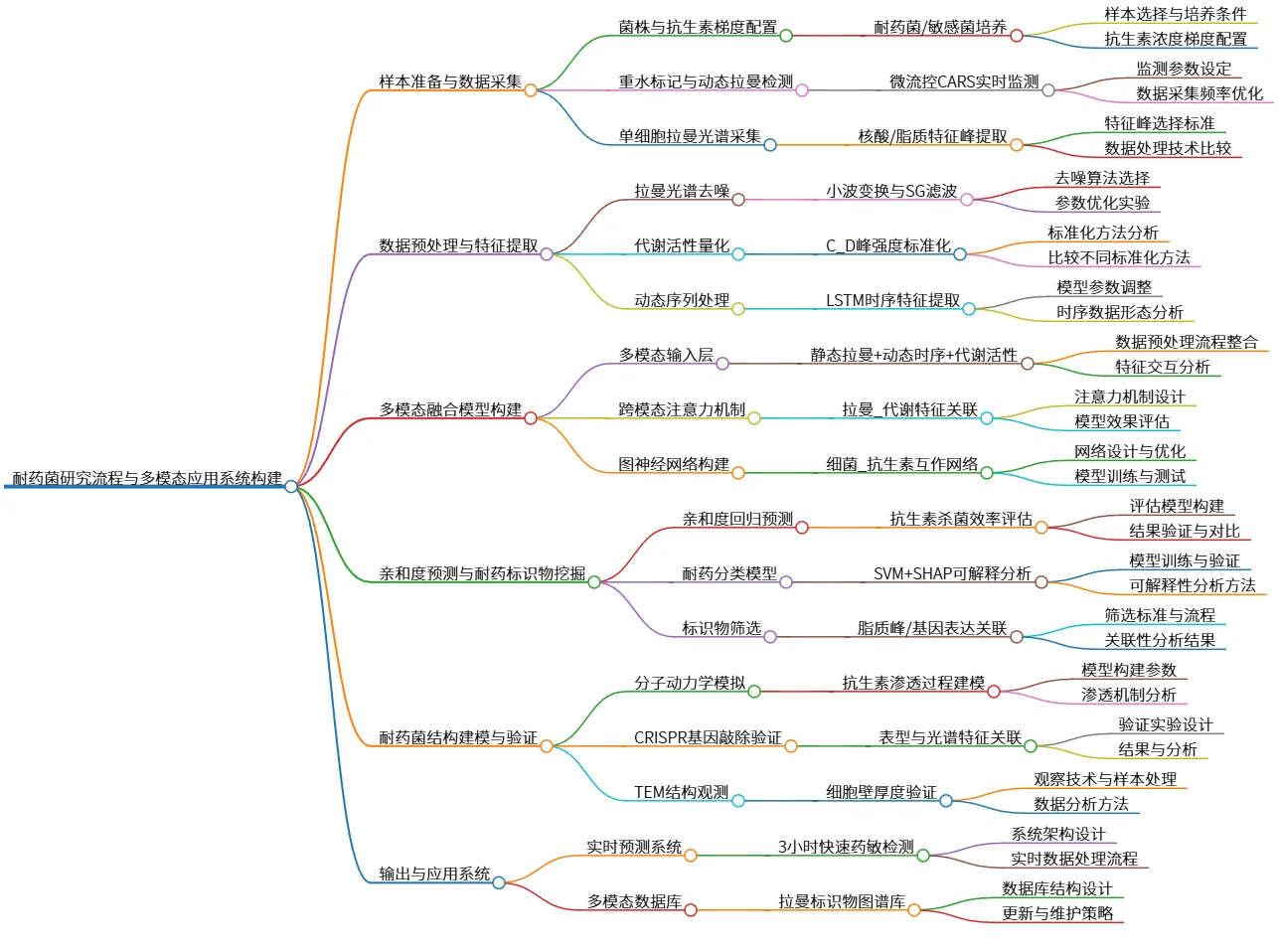
该思维导图详细描述了针对耐药菌的研究流程,包括样本准备与数据采集、数据预处理与特征提取、多模态融合模型构建、亲和度预测与耐药标识物挖掘、耐药菌结构建模与验证,以及最终的输出与应用系统。研究重点在于利用拉曼光谱、动态监测与机器学习技术,进行耐药细菌特征分析和预测,旨在提高抗生素的使用效率及耐药性识别的准确性。
源码
# 耐药菌研究流程与多模态应用系统构建
## 样本准备与数据采集
- 菌株与抗生素梯度配置
- 耐药菌/敏感菌培养
- 样本选择与培养条件
- 抗生素浓度梯度配置
- 重水标记与动态拉曼检测
- 微流控CARS实时监测
- 监测参数设定
- 数据采集频率优化
- 单细胞拉曼光谱采集
- 核酸/脂质特征峰提取
- 特征峰选择标准
- 数据处理技术比较
## 数据预处理与特征提取
- 拉曼光谱去噪
- 小波变换与SG滤波
- 去噪算法选择
- 参数优化实验
- 代谢活性量化
- C_D峰强度标准化
- 标准化方法分析
- 比较不同标准化方法
- 动态序列处理
- LSTM时序特征提取
- 模型参数调整
- 时序数据形态分析
## 多模态融合模型构建
- 多模态输入层
- 静态拉曼+动态时序+代谢活性
- 数据预处理流程整合
- 特征交互分析
- 跨模态注意力机制
- 拉曼_代谢特征关联
- 注意力机制设计
- 模型效果评估
- 图神经网络构建
- 细菌_抗生素互作网络
- 网络设计与优化
- 模型训练与测试
## 亲和度预测与耐药标识物挖掘
- 亲和度回归预测
- 抗生素杀菌效率评估
- 评估模型构建
- 结果验证与对比
- 耐药分类模型
- SVM+SHAP可解释分析
- 模型训练与验证
- 可解释性分析方法
- 标识物筛选
- 脂质峰/基因表达关联
- 筛选标准与流程
- 关联性分析结果
## 耐药菌结构建模与验证
- 分子动力学模拟
- 抗生素渗透过程建模
- 模型构建参数
- 渗透机制分析
- CRISPR基因敲除验证
- 表型与光谱特征关联
- 验证实验设计
- 结果与分析
- TEM结构观测
- 细胞壁厚度验证
- 观察技术与样本处理
- 数据分析方法
## 输出与应用系统
- 实时预测系统
- 3小时快速药敏检测
- 系统架构设计
- 实时数据处理流程
- 多模态数据库
- 拉曼标识物图谱库
- 数据库结构设计
- 更新与维护策略
图片